Specialavsnitt vecka 6 2026: Viruskaraktärisering influensa
Här redovisas resultaten från den genetiska karakterisering av influensavirus som hittills genomförts vid Folkhälsomyndigheten under säsongen 2025–2026.
Ett representativt urval av influensapositiva prover som mottagits från kliniska laboratorier eller samlats in genom sentinelövervakning analyseras vidare genom sekvensering vid Folkhälsomyndigheten. Från vecka 40, 2025, till vecka 2, 2026, har totalt 91 prover sekvenserats, varav 37 influensa A(H1N1)pdm09-, 49 influensa A(H3N2)- och fem influensa B/Victoria-virus. Genetisk grupp fastställdes för de prover där sekvenseringsresultatet för HA-genen godkänts. Samtliga karakteriseringsdata har rapporterats till databasen Global Initiative on Sharing All Influenza Data (GISAID) samt till ECDC via det europeiska övervakningssystemet (TESSy). Resultaten från den genetiska karakterisering som hittills genomförts denna säsong presenteras nedan.
Genetiska grupper - HA
Hemagglutinin-genen har vidare karakteriserats genom sekvensering för 37 influensa A(H1N1)pdm09-virus, 48 influensa A(H3N2)-virus och 5 influensa B/Victoria-virus. De resulterande genetiska grupperna visas i Tabell 1 samt i de fylogenetiska träden i bild 1–3. De antigena egenskaperna hos dessa virus har inte undersökts, men ett urval av dem har skickats till WHO:s samarbetscentrum (CC) i London för vidare analys. De övergripande antigena egenskaperna hos de genetiska grupper som presenteras i följande avsnitt baseras på sammanfattningen av antigena analyser som rapporterades vid det senaste WHO-mötet för vaccinsammansättning, som hölls i september 2025.
Influensa A(H1N1)pdm09
Av de karaktäriserade svenska influensa A(H1N1)pdm09-virusen tillhörde 36 stycken den genetiska gruppen 5a.2a.1, undergrupp D.3.1, representerad av A/Missouri/11/2025 och ett virus tillhörde genetisk grupp 5a.2a, undergrupp C.1.9.3, representerad av A/Hungary/286/2024 (se bild 1). Ingående vaccinvirus för subtyp A(H1N1)pdm09 för användning under norra halvklotets influensasäsong 2025–2026, A/Victoria/4897/2022 (äggodlat) och A/Wisconsin/67/2022 (cellodlat), tillhör båda genetisk grupp 5a.2a.1. Illerantisera framställda mot dessa vaccinvirus kände väl igen virus i både genetisk grupp 5a.2a och 5a.2a.1, medan humanserologiska studier har visat på mer skiftande resultat för aktuella cirkulerande virus. Det är därför svårt att i det här skedet dra en slutsats om vaccineffektiviteten för subtyp A(H1N1)pdm09 för norra halvklotets vaccin säsong 2025–2026.
Influensa A(H3N2)
Alla svenska A(H3N2)-virus som karaktäriserats för HA-genen tillhörde genetisk grupp 2a.3a.1. Av de 48 analyserade virusen tillhörde ett virus genetisk undergrupp J.2.2, representerad av A/Lisboa/216/2023, 7 tillhörde J.2.4 och 40 stycken tillhörde genetisk undergrupp J.2.4.1, även kallad K-varianten, representerad av A/Norway/8765/2025 (se bild 2). Undergrupp K definieras av aminosyrautbyte K2N, S144N, N185D, I160K, Q173R, T328A och S378N. Sju stycken stammar saknade följande tre substitutioner jämfört med undergrupp K: K2N, Q173R och S378N. Dessa sju stammar tillhör J.2.4 och är en ”intermediär grupp” till undergrupp K. Influensa A(H3N2)-vaccinvirus för användning under influensasäsongen 2025–2026 på norra halvklotet, A/District of Columbia/27/2023-liknande (cellodlat) och A/Croatia/10136RV/2023-liknande virus (äggodlat), tillhör båda genetisk grupp 2a.3a.1, undergrupp J.2. Analyser med illerantisera framställda mot dessa vaccinvirus samt humanserologiska analyser har båda visat på eventuell dålig match mellan virus inom genetisk undergrupp J.2.4 och norra halvklotets vaccinvirus för subtyp A(H3N2). Analyser för virus inom undergrupp K (J.2.4.1) pågår. I början av säsongen tydde tillgängliga data på att detta kunde potentiellt leda till en lägre vaccineffektivitet mot influensa A(H3N2) under säsongen, men preliminära resultat om vaccineffektivitet från England, Frankrike, VEBIS och Kanada visar att effektivitetsnivåerna är måttliga och jämförbara med säsongen 2024–2025.
- England: Early influenza virus characterisation and vaccine effectiveness in England in autumn 2025, a period dominated by influenza A(H3N2) subclade K (eurosurveillance.org)
- Europa: Early estimates of seasonal influenza vaccine effectiveness against influenza requiring medical attention at primary care level in Europe, week 41 - 49, 2025 (cdc.europa.eu)
- Kanada: Interim 2025/2026 influenza vaccine effectiveness estimates with immuno-epidemiological considerations for A(H3N2) subclade K protection, Canada, January 2026 (eurosurveillance.org)
- Frankrike: Interim vaccine effectiveness against influenza virus among outpatients, France, October 2025 to January 2026 (eurosurveillance.org)
Influensa B/Victoria
De fem B/Victoria-virus som karakteriserats i Sverige denna säsong tillhörde den genetiska gruppen V1A.3a.2 (C), representerad av B/Austria/1359417/2021 (se bild 3). Tre av de karakteriserade virusen tillhörde genetisk undergrupp C.5.6.1, representerad av B/ENG/120/2025, och två virus tillhörde genetisk undergrupp C.5.6, representerad av B/Switzerland/329/2024 (se tabell 1). Illerantisera framställda mot vaccinvirus för norra halvklotet 2025–2026, B/Austria/1359417/2021, kände väl igen virus i undergrupp C.5.6.1 samt C.5.6.
| Sub-/linjetyp | Genetisk grupp | Representerad av | Antal virus |
|---|---|---|---|
| A(H1N1)pdm09 | 5a.2a (C.1) | A/Netherlands/10468/2023 | 0 |
| A(H1N1)pdm09 | 5a.2a (C.1.9) | A/Lisboa/188/2023 | 0 |
| A(H1N1)pdm09 | 5a.2a (C.1.9.3) | A/Hungary/286/2024 | 1 |
| A(H1N1)pdm09 | 5a.2a.1 (D) | A/Victoria/4897/2022 | 0 |
| A(H1N1)pdm09 | 5a.2a.1 (D.3.1) | A/Missouri/11/2025 | 36 |
| A(H3N2) | 2a.3a.1 (J) | A/Thailand/8/2022 | 0 |
| A(H3N2) | 2a.3a.1 (J.2) | A/Croatia/10136RV/2023 | 0 |
| A(H3N2) | 2a.3a.1 (J.2.1) | A/Switzerland/59652/2024 | 0 |
| A(H3N2) | 2a.3a.1 (J.2.2) | A/Lisboa/216/2023 | 1 |
| A(H3N2) | 2a.3a.1 (J.2.3) | A/Netherlands/10685/2024 | 0 |
| A(H3N2) | 2a.3a.1 (J.2.4) | A/Singapore/GP20238/2024 | 7 |
| A(H3N2) | 2a.3a.1 (J.2.4.1) (K) | A/Norway/8765/2025 | 40 |
| A(H3N2) | 2a.3a.1 (J.2.5) | A/Victoria/211/2025 | 0 |
| B/Victoria | V1A.3a.2 (C) | B/Austria/1359417/2021 | 0 |
| B/Victoria | V1A.3a.2 (C.3.1) | B/Kanagawa/AC2414/2025 | 0 |
| B/Victoria | V1A.3a.2 (C.5) | B/Stockholm/3/2022 | 0 |
| B/Victoria | V1A.3a.2 (C.5.1) | B/Catalonia/2279261NS/2023 | 0 |
| B/Victoria | V1A.3a.2 (C.5.6) | B/Switzerland/329/2024 | 2 |
| B/Victoria | V1A.3a.2 (C.5.6.1) | B/ENG/120/2025 | 3 |
| B/Victoria | V1A.3a.2 (C.5.7) | B/Greece/5509/2024 | 0 |
| B/Yamagata | Y3 | 0 |
Analys av antiviral resistens – NA, PA och M
Inget av de 35 influensa A(H1N1)pdm09-, 48 influensa A(H3N2)- eller fem influensa B/Victoria-virus som analyserades genetiskt avseende känslighet för neuraminidashämmare (NAI) bar någon av de aminosyrasubstitutioner i NA-genen som är kända för att ge nedsatt eller mycket nedsatt känslighet mot neuraminidashämmarna oseltamivir eller zanamivir.
PA-genen hos 33 influensa A(H1N1)pdm09-, 47 influensa A(H3N2)- och fem influensa B/Victoria-virus analyserades genetiskt avseende känslighet för PA-hämmaren (PAI) baloxavir. Inga substitutioner kopplade till baloxavirresistens påvisades.
Matrixgenen analyserades hos 37 influensa A(H1N1)pdm09- och 49 influensa A(H3N2)-virus och samtliga bar substitutionen S31N, som ger resistens mot amantadin.
Bild 1. Fylogenetiskt träd för influensa A(H1N1)pdm09 hemagglutinin (HA1), aminosyra. Källa: GISAID.
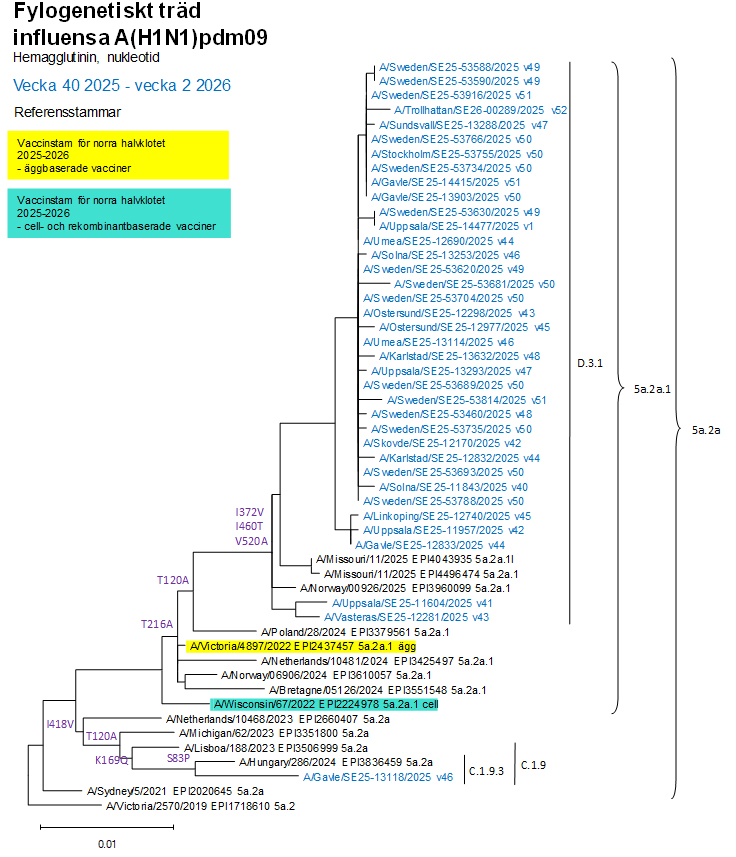
Se bild för Fylogenetiskt träd för influensa A(H1) i större format
Bild 2. Fylogenetiskt träd för influensa A(H3N2) hemagglutinin (HA1), aminosyra. Källa: GISAID.
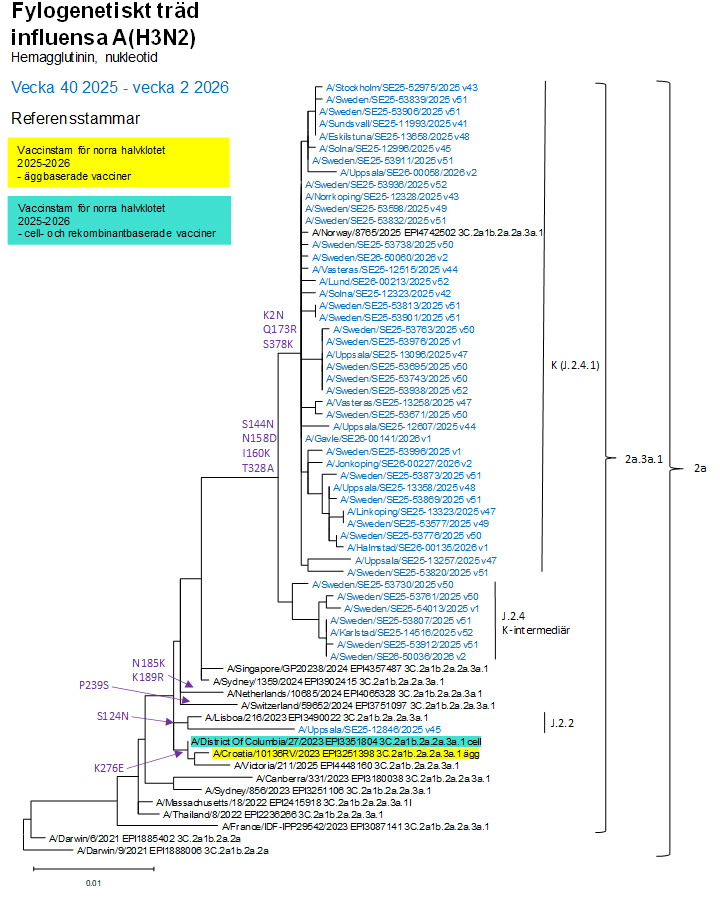
Se bild för fylogenetiskt träd för influensa A(H3N2) i större format
Bild 3. Fylogenetiskt träd för influensa B/Victoria hemagglutinin (HA1), aminosyra. Källa: GISAID.
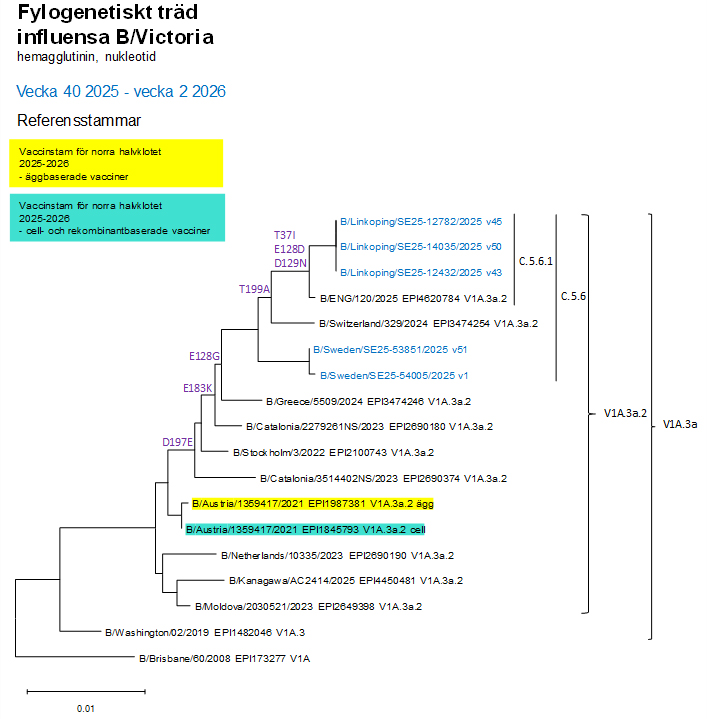
Se bild för fylogenetiskt träd för B/Victoria i större format
