Gemensam nationell hantering av sekvenseringsdata inom klinisk mikrobiologi (GENSAM)
LyssnaGENSAM är Folkhälsomyndighetens plattform och databas för automatiserad analys av helgenomsekvenseringsdata.
Landets kliniska mikrobiologiska laboratorier som utför sekvenseringen lokalt deponerar genererade data som sedan analyseras centralt och delas med smittskyddsenheter samt berörda aktörer. I dagsläget hanteras SARS-CoV-2 av GENSAM men även andra smittämnen är under implementation. GENSAM har delfinansierats med hjälp av Vinnova och Europeiska kommissionen.
För frågor kring GENSAM skicka e-post till gensam@folkhalsomyndigheten.se.
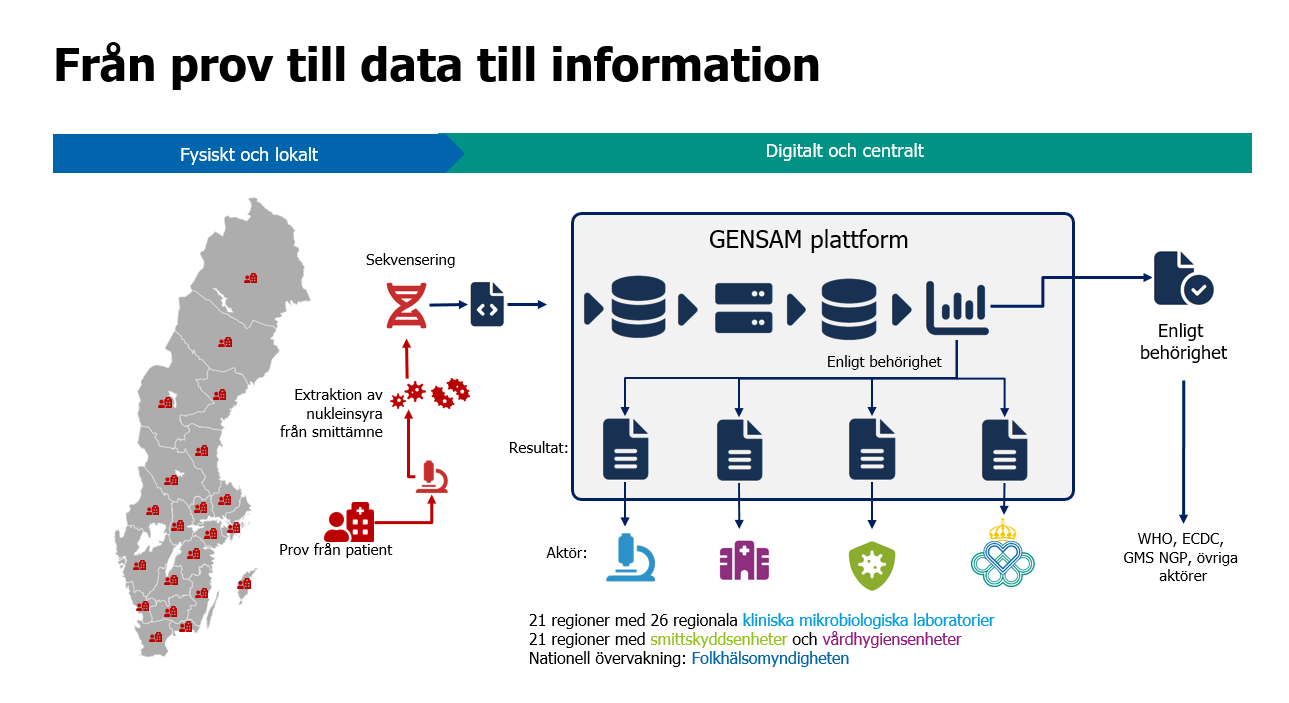
Figur 1. Från prov till data till information.
Symboler från Font Awesome Pro CC-BY 4.0 License (fontawesome.com)
Patientprov förvandlas från data till information genom nukleinsyraextraktion och sekvensering av smittämnen. GENSAM-plattformen analyserar sekvensdata och delar resultaten med berörda aktörer. Berörda aktörer är 21 regioner med 26 regionala kliniska mikrobiologiska laboratorier, 21 regioner med smittskyddsenheter och vårdhygiensenheter samt Nationell övervakning: Folkhälsomyndigheten.

